Hace miles de millones de años, antes de que existieran las bestias, las bacterias o cualquier organismo vivo, existían los RNA. Estas moléculas probablemente se arremolinaban con aminoácidos y otras biomoléculas rudimentarias, fusionándose y divergiendo, en un crisol de un planeta que de otro modo estaría sin vida.
Entonces, de alguna manera, surgió algo especial: una máquina simple, un bolsillo hecho de RNA, con la capacidad de colocar aminoácidos uno al lado del otro y tal vez unirlos en cadenas. Esta fue la macromolécula que evolucionaría gradualmente hacia el ribosoma, el complejo RNA-proteína responsable de traducir la información genética en proteínas. Su nacimiento, cuyos detalles siguen siendo hipotéticos, habría creado un cambio fundamental en este mundo prebiótico dominado por el RNA, proporcionando un ingrediente clave para toda la vida tal como la conocemos. Ada Yonath, bióloga estructural del Instituto de Ciencias Weizmann en Rehovot, Israel, y su equipo, conceptualizaron por primera vez la idea de ‘protoribosoma’ hace casi dos décadas, después de que ella y otros determinaran la estructura del ribosoma moderno, una hazaña que luego aseguró a Yonath ,una parte del Premio Nobel de Química 2009.
=> Recibir por Whatsapp las noticias destacadas
Pero para solidificar el caso del protoribosoma hipotético, Yonath y su laboratorio tendrían que construirlo.
Y los logros del laboratorio en los últimos dos años (la creación de una máquina de RNA primitiva que puede unir dos aminoácidos entre sí) (1,2) han creado una oleada de entusiasmo. Un grupo en Japón, trabajando por separado y dirigido por el biólogo molecular Koji Tamura en la Universidad de Ciencias de Tokio, logró crear un protoribosoma(3) funcional similar.
El trabajo de Yonath y Tamura parece recapitular un hito en el camino desde las moléculas orgánicas primordiales hasta el ribosoma utilizado por el último ancestro común de todos los seres vivos. No fué una tarea sencilla: en el grupo de Yonath, el proyecto pasó de un investigador a otro y tardó más de 15 años en tener éxito. El trabajo ahora ha abierto la puerta para que los científicos del origen de la vida completen más detalles. Y otros están considerando el protoribosoma, o algo parecido, como una herramienta para crear nuevos tipos de biomoléculas.
Este debería ser un punto de partida para muchos más campos de investigación.
Los científicos han estado intentando recrear alguna apariencia del origen químico de las biomoléculas durante décadas; Hace 70 años, Stanley Miller, químico de la Universidad de Chicago en Illinois, provocó una mezcla de gases para crear compuestos orgánicos( 4). Investigadores como Carl Woese y Francis Crick sugirieron que el ribosoma podría haber comenzado como una molécula hecha únicamente de RNA, una idea que fue respaldada hace 40 años con la prueba de que los RNA pueden catalizar reacciones(5,6). Esto condujo a la hipótesis del ‘mundo del RNA’, que describe un tiempo, antes de las células o de la vida real, cuando los RNA se replicaban y catalizaban reacciones (7). La idea del mundo del RNA se ha puesto en duda en la última década; muchos científicos ahora sospechan que una variedad de biomoléculas, incluidas proteínas rudimentarias, lípidos y metabolitos, podrían haber existido junto con los ácidos nucleicos.
Yonath sospecha que había muchos RNA en la tierra primitiva. La mayoría de ellos ya no existen porque eran pequeños y no útiles. El protoribosoma, fué el que perduró.
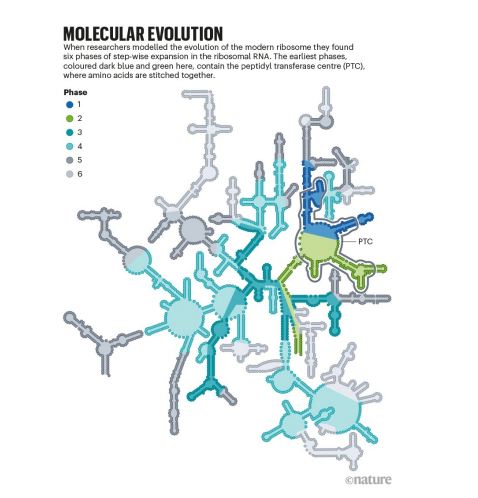
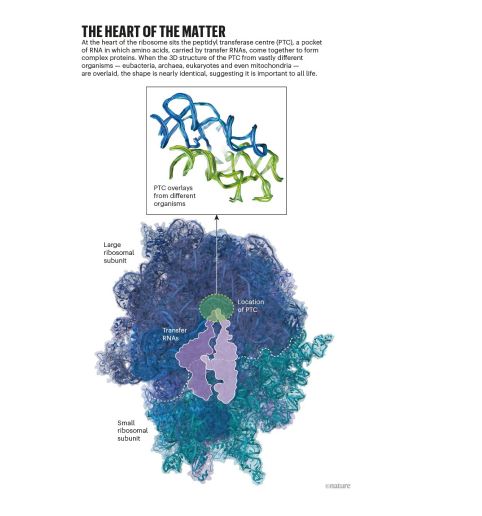
Yonath y sus colegas publicaron estructuras de alta resolución para las dos subunidades de proteína-RNA que forman un ribosoma ,a principios de la década de 20008,(9). Estos ejemplos provienen de bacterias extremófilas. Y a medida que se publicaban las estructuras ribosómicas de otros organismos, en lo profundo del núcleo de la subunidad grande , había un segmento semisimétrico. Esta región contenía una estructura similar a un bolsillo, hecha de RNA ribosómico, llamada centro de peptidil transferasa (PTC). Durante la traducción del mRNA en proteína, cuando se colocan dos aminoácidos en el PTC, se crean las condiciones para que se unan. Y, aunque la secuencia específica de nucleótidos de la estructura varió entre las especies, la forma fué la misma en todos los ejemplos, lo que sugiere que es crucial para la capacidad del ribosoma para sustentar la vida .
En 2006, Yonath y su equipo decidieron estudiar de cerca sobre la evolución del ribosoma, particularmente observar de cerca la región semisimétrica que contiene el PTC. Especularon que este es el protoribosoma, esta es la parte de la que evolucionó el ribosoma. Pero, la región que estaban postulando como el protoribosoma putativo se ensambla a partir de 178 ribonucleótidos, que sería una estructura terriblemente grande para surgir, completamente formada, en la tierra primordial. Basado en parte en la simetría que había observado, Agmon propuso un modelo que requería 2 RNA similares en forma de L de 60 y 61 nucleótidos. El equipo pensó que este era un tamaño de molécula más razonable para surgir en la tierra primitiva.
Los fragmentos de ese tamaño son ciertamente plausibles. Elisa Biondi, bioquímica de la Fundación para la Evolución Molecular Aplicada en Alachua, Florida, y sus colegas lograron sintetizar RNA de aproximadamente 100 a 300 bases de longitud en materiales llamados cristales de roca, que se habrían formado debido a la acción volcánica o impactos de meteoritos en un mundo prebiótico(10).
Pero no todo el mundo está convencido de que los fragmentos cortos de Yonath y Agmon surgieran espontáneamente. Joseph Moran, un químico orgánico de la Universidad de Estrasburgo en Francia, elogia los logros de Yonath, pero duda de que el protoribosoma simplemente haya aparecido. Tenía que derivarse de cosas mucho más simples, arguye.
Robert Root-Bernstein, biólogo de la Universidad Estatal de Michigan en East Lansing, tiene una teoría sobre lo que podrían haber sido esas cosas más simples: RNA de transferencia (tARN). En el ribosoma moderno, los aminoácidos ingresan al PTC unidos a tRNA que coinciden con los códigos de tres letras (llamados cordones) en el mRNA. Esos códigos determinan qué aminoácido viene después en la proteína. Para Root-Bernstein y otros, el núcleo de PTC se parece mucho a cuatro tRNA unidos. Y los tRNA, señala, no solo entregan aminoácidos al ribosoma; son moléculas versátiles que pueden realizar todo tipo de tareas, como detectar nutrientes y silenciar genes. Quizás tenían alguna función antes del protoribosoma, y luego proporcionaron los componentes básicos para esa estructura.
Independientemente de cómo surgió el protoribosoma hipotético, en el momento en que Agmon y Yonath lo concibieron por primera vez, no había evidencia experimental que demostrara que podría haber existido y funcionado como pensaban. La hipótesis del protorribosoma asume que uno de esos primeros bolsillos de RNA podría unir los aminoácidos y que luego evolucionó hasta convertirse en el ribosoma actual, que tiene la misma función. Esto es una buena idea, pero no es un hecho.
El DNA más antiguo muestra que los mastodontes vagaban por Groenlandia hace 2 millones de años.
Las teorías audaces requieren evidencia extraordinaria, y eso es lo que el laboratorio de Yanoth se propuso obtener. El primer paso, fué producir las moléculas para construir este protoribosoma hipotético
Davidovich estudió las secuencias de RNA de varios ribosomas modernos. La macromolécula contiene docenas de ribonucleótidos y proteínas accesorias, pero no todos están involucrados en la forma o las funciones del PTC. Eliminó todo lo que parecía ser extraño al protoribosoma, dejando suficiente RNA para crear ese bolsillo semisimétrico(2). Algunos de estos RNA pudieron emparejarse en algo similar al núcleo de PTC que Agmon había imaginado.
El segundo paso fué demostrar que estos supuestos protorribosomas podían tomar dos aminoácidos y unirlos.
Davidovich intentó mostrar la actividad de PTC de la misma manera que otros investigadores midieron la capacidad del ribosoma moderno para unir dos análogos de aminoácidos en un dipéptido. Etiquetó tales análogos, vinculados a un puñado de nucleótidos para sustituir al tRNA, con radioctividad. Después de mezclarlos con el protoribosoma, pensó que sería capaz de clasificar las moléculas por tamaño y encontrar dipéptidos radiomarcados más largos. Pero nunca vio una pizca de dipéptido.
La estudiante graduada Miri Krupkin, quién usó las construcciones de Davidovich y otras que diseñó ella misma, también intentó detectar el producto de dipéptido radiactivo en función de su tamaño, intentó marcar los sustratos con fluorescencia, en lugar de radioactividad. Los resultados fueron negativos.
Krupkin comenzó a preguntarse si el protoribosoma incluso producía péptidos cuando apareció por primera vez. Probó otras químicas potenciales, como agregar o eliminar grupos fosfato de otras moléculas, y aun así no obtuvo resultados.
La teoría del origen de la vida que implica un híbrido RNA–proteína obtiene nuevo apoyo
Bose, que se incorporó al laboratorio de Yonath como postdoctorado en 2016, llevaría el proyecto hasta el final. Se había formado como química y eso le dió una nueva perspectiva. Sabía que si el protoribosoma primitivo funcionaba, probablemente sería ineficaz y produciría una pequeña cantidad de dipéptido. En lugar de separar los productos de reacción por tamaño, Bose recurrió a la espectrometría de masas, que había avanzado hasta convertirse en el método más sensible en ese momento. Todavía no fué fácil: realizó muchas reacciones y experimentos de control, y probó dos formas de espectrometría de masas, pero finalmente vió un pico que representaba el dipéptido esperado!!! (1,2).
La cantidad de producto era minúscula. Ella sospecha que Davidovich y Krupkin podrían haberlo estado haciendo todo el tiempo, pero no había suficiente para detectarlo usando sus métodos.
Cuando Krupkin estaba tratando de medir un producto de dipéptido, el grupo de Yonath no era el único que seguía el rastro de la máquina mínima de unión de péptidos. Tamura, en Japón, también se inspiró en el bolsillo central semisimétrico. Reclutó a su grupo a la estudiante de maestría Mai Kawabata y al estudiante universitario Kentaro Kawashima para crear su propia estructura similar a un protorribosoma.
Su facsímil de PTC, hecho de dos RNA de 74 nucleótidos, era similar al de Yonath, pero para sustituir a los tRNA modernos, utilizaron estructuras llamadas minihélices. Estos son aproximadamente la mitad del tamaño de un tRNA moderno y, por lo tanto, eran considerablemente más grandes que los reemplazos de tRNA que usó Yonath. Se cree que los tRNA modernos evolucionaron a partir de minihélices.
El equipo de Tamura finalmente logró su propio éxito, detectando también el dipéptido con espectrometría de masas(3).
Las minihélices fueron una adición clave. Lo llevó al siguiente nivel, por así decirlo, en el sentido de que construyó lo que muchos evolucionistas creen que es el tRNA primordial. Algunos científicos, incluso sospechan que las minihélices desarrollaron la capacidad de autorreplicarse, otro paso clave en el camino hacia los organismos vivos.
Tamura advierte que ninguna de las construcciones del laboratorio funciona exactamente como lo hace el PTC moderno. Todos nuestros resultados son demasiado simples, dice. «Todavía tenemos un largo camino por recorrer antes de que realmente entendamos la evolución del PTC y el ribosoma».
Lo que tiene de especial el trabajo del protoribosoma, es que es posible imaginar cómo este núcleo primitivo, durante milenios, podría haber acumulado piezas adicionales de RNA y proteína para crear el ribosoma moderno.
Petrov y sus colegas han predicho exactamente ese tipo de línea de tiempo trabajando hacia atrás desde el ribosoma actual. Analizaron los ribosomas en busca de «huellas dactilares» a nivel atómico que quedaron en la estructura del ribosoma 3D cuando se agregaron nuevas ramas de RNA(13). Esto condujo a un modelo que mostraba estructuras tempranas que coincidían con las del protoribosoma de Yonath, con piezas adicionales de RNA incorporadas lentamente con el tiempo .
Un próximo paso obvio para el equipo de Yonath es tratar de hacer un conjunto diverso de péptidos que tengan más de dos aminoácidos.
Hay un grupo que afirma haber sintetizado un péptido más largo a partir de una estructura similar a un protorribosoma. Yuhong Wang, biofísica de la Universidad de Houston en Texas, y su colega informaron resultados de espectrometría de masas de experimentos con una versión más grande del PTC que indican la producción de una cadena de nueve lisinas (14). Wang cree que también podría haber polímeros de lisina de otras longitudes.
Wang y otros están interesados en usar esos ribosomas reducidos para fabricar nuevos tipos de biomoléculas, no limitadas a los 20 aminoácidos habituales o incluso a los aminoácidos, que podrían tener usos en la medicina o la industria. Por ejemplo, podrían construir moléculas a partir de aminoácidos que tienen una estructura levógira, a diferencia de los levógiros que se ven en la vida en la tierra. Este tipo de síntesis de macromoléculas podría ser más barata y más respetuosa con el medio ambiente que otros medios.
Mientras tanto, queda mucha investigación por hacer para los estudios del origen de la vida. Los científicos necesitan averiguar cómo los RNA adquirieron la capacidad de autorreplicarse. Y necesitan descubrir cómo un ribosoma primitivo habría logrado crear péptidos específicos codificados por mRNA primitivos. Esos procesos, más la capacidad de sintetizar péptidos, proporcionarían la materia prima para la evolución.
Hay un factor más : los primeros péptidos producidos por el protoribosoma deben haber sido útiles de alguna manera, o no habría ninguna ventaja evolutiva para la existencia continua de la máquina. Esto sugiere un par de funciones especulativas: tal vez los péptidos secuestraron iones metálicos que de otro modo destruirían los RNA. O bien, podrían haber ayudado a formar compartimentos biomoleculares tempranos para concentrar el RNA y los péptidos juntos.
Cuando obtienes algo sobre lo que la evolución puede actuar , el resto es historia.
Ronald Palacios Castrillo, M.D.,PhD.
Referencias Bibliográficas
1.Bose, T., Fridkin, G., Bashan, A. & Yonath, A. Israel J. Chem. 61, 863–872 (2021).
2.Bose, T. et al. Nucleic Acids Res. 50, 1815–1828 (2022).
3. Kawabata, M. et al. Life 12, 573 (2022).
4. Miller, S. L. Science 117, 528–529 (1953).
5. Kruger, K. et al. Cell 31, 147–157 (1982).
6. Guerrier-Takada, C., et.al.,. Cell 35, 849–857 (1983).
7. Gilbert, W. Nature 319, 618 (1986).
8. Schluenzen, F. et al. Cell 102, 615–623 (2000).
9. Harms, J. et al. Cell 107, 679–688 (2001).
10. Jerome, C. A.,et.al., Astrobiology 22, 629–636 (2022).
11. Forsythe, J. G. et al. Angew. Chem. Int. Edn Engl. 54, 9871–9875 (2015).
12. Müller, F. et al. Nature 605, 279–284 (2022).
13. Petrov, A. S. et al. Proc. Natl Acad. Sci. USA 111,10251–10256 (2014).
14. Xu, D. & Wang, Y. Biochem. Biophys. Res. Commun. 544, 81–85 (2021).