Ronald Palacios Castrillo
Resumen
En esta trabajo Jones,et.al.(Cell. https://doi.org/10.1016/j.cell.2024.03.045 ) revisa la actualidad del sistema inmunológico de las plantas.
Las enfermedades de las plantas causan hambrunas, impulsan la migración humana y presentan desafíos para la sostenibilidad agrícola a medida que los rangos de patógenos cambian debido al cambio climático. Los fitomejoradores descubrieron loci genéticos mendelianos que confieren resistencia a enfermedades a aislados de patógenos específicos hace más de 100 años.
El mejoramiento posterior para lograr resistencia a las enfermedades sustenta la agricultura moderna y, junto con la aparición y el enfoque en plantas modelo para la investigación genética y genómica, ha proporcionado ricos recursos para la exploración de la biología molecular durante los últimos 50 años.
Estos estudios condujeron a la identificación de receptores extracelulares e intracelulares que convierten el reconocimiento de patrones moleculares codificados por microbios extracelulares o efectores de virulencia intracelulares administrados por patógenos en activación de defensa.
Estos sistemas receptores y las respuestas posteriores definen los sistemas inmunológicos de las plantas que han evolucionado desde la migración de las plantas a la tierra hace aproximadamente 500 millones de años.
Nuestro conocimiento actual de los sistemas inmunológicos de las plantas proporciona la plataforma para el desarrollo de una mejora racional de la resistencia para controlar las numerosas enfermedades que siguen plagando la producción de cultivos.
Las enfermedades de las plantas impactan la salud humana y ambiental
En 1974, los mecanismos que sustentan las interacciones entre plantas y microbios no se entendían bien, pero claramente se necesitaba una mejor comprensión para permitir un control eficaz de las enfermedades de los cultivos.
Las pandemias de enfermedades vegetales han afectado a las sociedades y a la seguridad alimentaria desde la antigüedad.
Entre muchos ejemplos, el tizón tardío de la papa, causado por el oomiceto Phytophthora infestans, provocó la hambruna irlandesa de la papa en la década de 1840, lo que impulsó la emigración de millones de personas que dieron como resultado una gran comunidad irlandesa en los Estados Unidos.
Los hábitos ingleses de consumo de té son consecuencia de una epidemia de roya fúngica que destruyó los cultivos de café de Sri Lanka, lo que obligó a cambiar al té.
La roya del tallo transmitida por el viento puede devastar los rendimientos del trigo y en América del Norte se produjeron enormes pérdidas de cereales en 1903 y 1905 y entre 1950 y 1954. A pesar de muchos avances desde 1974, las enfermedades de los cultivos no han desaparecido.
La producción de naranjas de Florida ha caído más del 80% en los últimos cinco años, impulsada por una enfermedad transmitida por insectos causada por la bacteria Candidatus liberibacter.
El cacao (un cultivo arbóreo y fuente del chocolate), el café, la yuca y el plátano se ven afectados por enfermedades fúngicas, oomicetos, bacterianas y virales mal controladas que reducen en gran medida el rendimiento y aumentan el costo del cultivo.
Estas llamadas de atención alentaron el reclutamiento en la década de 1970 en el campo emergente de las interacciones moleculares entre plantas y microbios (MPMI).
En 2019 se aplicaron aproximadamente 2 millones de toneladas de agroquímicos en la producción agrícola, de los cuales el 47,5% fueron herbicidas, el 29,5% insecticidas y el 17,5% fungicidas. Sólo en fungicidas se gastan más de 20.000 millones de dólares al año.
El cambio climático está exacerbando los desafíos de la producción de cultivos, incluido el cambio en los rangos de crecimiento de patógenos en todo el mundo. Sin embargo, las soluciones genéticas siguen siendo controvertidas en muchas jurisdicciones, a pesar de su obvia preferencia a las soluciones químicas.
El descubrimiento biológico está impulsado por elecciones juiciosas de sistemas modelo. Un desafío duradero para los investigadores del MPMI ha sido la diversidad de patógenos (hongos, bacterias, oomicetos, virus o invertebrados) y especies de plantas hospedantes.
Tradicionalmente, la economía impulsó la financiación para apoyar estudios sobre enfermedades con mayor impacto en el rendimiento de los cultivos, incluso si eran menos adecuados para revelar conocimientos biológicos novedosos.
Las primeras investigaciones del MPMI también priorizaron las interacciones simbióticas entre las leguminosas y el simbionte bacteriano fijador de nitrógeno Rhizobium y el mecanismo por el cual Agrobacterium, que forma agallas de la corona (un pariente cercano de Rhizobium), entrega DNA a las células vegetales.
Esto probablemente se debió a que las técnicas moleculares de las décadas de 1970 y 1980 permitieron descubrimientos con bacterias, pero menos con plantas.
En la primera reunión del MPMI en Bielefeld, Alemania, en 1982, casi todas las presentaciones versaron sobre Rhizobium o Agrobacterium, y sólo una de las seis sesiones versó sobre los mecanismos de virulencia de los patógenos bacterianos Pseudomonas, Xanthomonas y Erwinia. No hubo sesiones sobre patógenos eucariotas ni sobre mecanismos moleculares de resistencia a las enfermedades.
Estado del arte en inmunidad vegetal desde antes de 1974 hasta 1994
Los análisis genéticos de la resistencia a las enfermedades de las plantas comenzaron poco después del redescubrimiento de las leyes de herencia de Mendel con el informe de que la resistencia a los hongos patógenos de la roya en el trigo era conferida por genes únicos semidominantes de resistencia a enfermedades (R).
El fenotipo de resistencia o susceptibilidad a la enfermedad dependía no sólo del huésped sino también del genotipo del patógeno; algunas, pero no todas, razas de un hongo de la roya del trigo podrían superar genes R particulares y causar enfermedades.
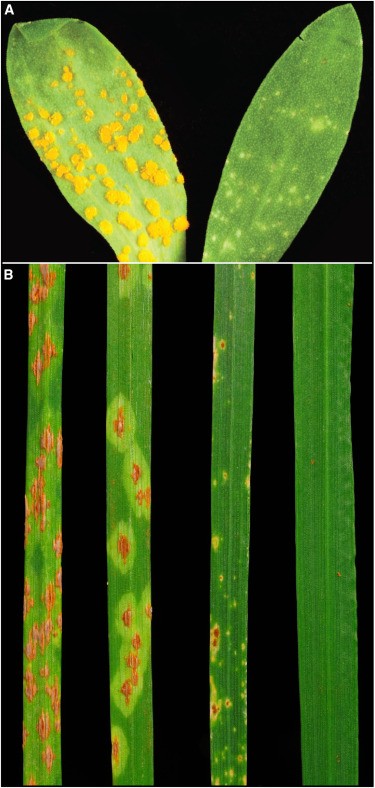
En estudios genéticos sobre la interacción del lino (Linum usitatissimum) y su hongo de la roya Melampsora lini, (Figura 1), H.H. Flor investigó la herencia de la capacidad de una raza de roya para ser resistida por un gen R particular y demostró que la “reconocibilidad” ( “Avirulencia”) es conferida por un gen dominante en el patógeno que fue emparejado por un gen R dominante correspondiente a esa raza en el huésped.
El concepto resultante se conoció como el modelo “gen por gen”, según el cual El gen de resistencia a enfermedades confiere el reconocimiento de un patógeno que porta un gen de avirulencia (Avr) correspondiente.
Esto, por supuesto, plantea la pregunta: ¿por qué un patógeno codificaría un producto que permite resistirlo? La aparente paradoja ya se ha resuelto; Los genes Avr del patógeno pueden contribuir a la virulencia del patógeno, siempre que el huésped no haya desarrollado un receptor inmunológico que lo detecte (detallado a continuación).
Los patógenos normalmente codifican y despliegan en las células vegetales cientos de estos factores de virulencia, generalmente denominados «efectores» de virulencia. Sin embargo, si uno de estos es reconocido por un receptor inmunológico del huésped, generalmente se activan defensas suficientes para frustrar el patógeno.
Los patógenos necesitan desplegar suficientes efectores de virulencia funcionalmente redundantes para infectar a un huésped, pero los huéspedes sólo necesitan detectar un efector para ser resistentes.
En poblaciones de huéspedes genéticamente diversas, cada individuo tiene diferentes conjuntos de capacidades de detección y la selección dependiente de la frecuencia limita el éxito de los patógenos, ya que una raza de patógenos que evade la detección en una planta de la población puede no poder hacerlo en la mayoría de las plantas.
Paralelamente, otra línea de investigación temprana identificó moléculas derivadas de patógenos (“elicitores”) que activan la defensa y definen el complejo conjunto de respuestas de defensa de las plantas que activan. Los elicitores se definieron inicialmente como moléculas patógenas que activan la biosíntesis de fitoalexinas, nombre asignado a un conjunto diverso de metabolitos secundarios antimicrobianos, muchos de los cuales derivan de la fenilalanina o el triptófano.
Esto impulsó investigaciones sobre el gen de la fenilalanina liasa (PAL) y su activación en varios sistemas. En particular, en cultivos celulares de perejil, un inductor secretado por Phytophthora megasperma conduce a la inducción transcripcional de PAL.
Estas investigaciones culminaron en un artículo histórico de Cell que define un péptido mínimo de 13 aminoácidos (Pep13) derivado de este inductor de transglutaminasa, su unión de alta afinidad a un presunto receptor en cultivos de células de perejil y el repertorio resultante de respuestas de defensa provocadas.
Además de la acumulación de fitoalexinas, la provocación también desencadena la producción de especies reactivas de oxígeno (ROS) a través de la NADPH oxidasa de la membrana plasmática, afluencias de Ca2+, activación de proteínas quinasas activadas por mitógenos (MAP) y otras proteínas quinasas, fortificación de la pared celular y genes relacionados con la activación de la defensa.
Las respuestas de defensa de este tipo, dirigidas al reconocimiento de patrones microbianos, llegaron a conocerse como inmunidad activada por patrones (PTI).
El marco conceptual de PTI fue iniciado por el fallecido Charles Janeway, Jr., quien sugirió que los patrones de patógenos relativamente conservados, como la flagelina o los lipopoisacáridos, eran estimulantes inmunes de metazoos específicos.
Esto, señaló, era la base probable del “pequeño secreto sucio de la inmunología” de que las respuestas inmunes a los antígenos inyectados a menudo requerían una mezcla de productos bacterianos conocidos como “adyuvantes”.
Polly Matzinger articuló un concepto contrastante, pero no mutuamente excluyente, quien sugirió que la diferenciación del yo del no-yo implicaba el reconocimiento de componentes «dañados» del huésped que ella denominó «señales de peligro».
Ahora aceptamos que los autocomponentes modificados derivados de células vegetales y las señales de peligro genéricas, como las especies reactivas de oxígeno, también desempeñan funciones clave en el sistema inmunológico de las plantas. Los primeros intentos de consolidar el pensamiento sobre los sistemas inmunológicos de metazoos y plantas fueron fundamentales para impulsar el campo MPMI.
Un tercer hilo paralelo implicó la investigación de fenotipos mutantes en los que la defensa parece activarse en ausencia aparente de cualquier patógeno.
Tales mutantes que imitan lesiones, a veces denominados “plantas paranoicas” (que activan la defensa incluso cuando no están siendo atacadas) pueden resultar de mutaciones recesivas en reguladores negativos de la defensa, o de mutaciones semidominantes en las que ha surgido un alelo de un receptor inmunológico, a través de una mutación. o cruce desigual en un locus que porta múltiples parálogos de un gen de receptor inmunológico, que es constitutivamente activo. Un ejemplo temprano clásico de esto último es el alelo Rp1-D21 del locus Rp1 del maíz que confiere resistencia al patógeno de la roya Puccinia sorghi.
Revisando las reseñas
Nuestra comprensión actual del MPMI y de la inmunidad de las plantas habría sido inimaginable en 1974. El modelo gen por gen fue clave para proporcionar un marco conceptual para comenzar el aislamiento de genes utilizando varios métodos que surgieron en la década de 1980.
Un enfoque en los sistemas modelo emergentes dio como resultado la clonación de genes de receptores inmunes y genes efectores, como se detalla a continuación.
El descubrimiento de que la mayoría de los genes R codifican proteínas repetidas ricas en leucina (NLR) de unión a nucleótidos proporcionó un avance crucial, como se detalla a continuación. Por el contrario, los genes efectores codifican proteínas con diversas funciones bioquímicas.
Se definieron mecanismos de reconocimiento directo o indirecto para los receptores inmunes de la superficie celular y los NLR, al igual que la interacción de los mecanismos de defensa iniciados por la unión de ligandos microbianos a los receptores de la superficie celular y la activación de los receptores NLR intracelulares.
La genómica aceleró el descubrimiento y análisis tanto de los repertorios de receptores inmunes de las plantas como de los complementos efectores de patógenos. Los avances importantes fueron acompañados por revisiones influyentes.,,,,,,,,,, El campo se ha vuelto demasiado vasto para una descripción completa de cada descubrimiento, por lo que intentamos resaltar los avances conceptuales más significativos en nuestro campo de los últimos 50 años. . En la Figura 2 se muestra una cronología de avances importantes.
Agrobacterium, tumores vegetales y el plásmido inductor de tumores (Ti) como máquina de virulencia y herramienta de investigación e ingeniería.
La forma en que la bacteria gramnegativa del suelo Agrobacterium tumefaciens causa enfermedades en las plantas y permite la transferencia de genes al DNA cromosómico de las plantas proporciona un ejemplo espectacular de cómo la ciencia impulsada por la curiosidad puede permitir una tecnología revolucionaria.
Los estrechamente relacionados A.tumefaciens y A. rhizogenes causan enfermedades de agallas de la corona y raíces peludas similares a tumores, respectivamente, en muchas plantas.
En un artículo histórico publicado en Cell, Chilton et al. demostraron que parte del plásmido inductor de tumores (Ti) de Agrobacterium se incorpora al DNA genómico del huésped.
El DNA transferido (T-DNA) transporta genes que codifican enzimas involucradas en la síntesis de hormonas de crecimiento de las plantas y para la síntesis de opiniones (conjugados de aminoácidos y azúcar) que las Agrobacterias pueden utilizar como fuente de carbono y nitrógeno.
Comprender la transferencia de DNA-T de Agrobacterium a las plantas fue un triunfo temprano para el campo del MPMI y un logro pionero que sustenta la modificación genética de los cultivos y creó una herramienta indispensable para investigar la función de los genes de las plantas.
El proceso de transferencia de DNA-T evolucionó a partir de un sistema de secreción tipo 4 involucrado en la conjugación bacteriana y el DNA-T se integra en el DNA del huésped en ubicaciones aleatorias, un descubrimiento que permitió la generación de colecciones de mutantes de saturación en la planta de referencia Arabidopsis thaliana.
Infiltración de agrobacterias
Las cepas que introducen genes de interés en hojas de plantas susceptibles, como Nicotiana benthamiana, dan como resultado una expresión genética transitoria que facilita enormemente las investigaciones de los mecanismos de defensa, las interacciones proteína-proteína y la preparación de proteínas para la biología estructural en los receptores inmunes de las plantas.
«¡Arabidopsis no tiene patógenos!» y los primeros días de la efectoronomía bacteriana
El modelo gen por gen y su predicción contraintuitiva de genes de «reconocibilidad» en patógenos impulsaron una búsqueda de genes de avirulencia (Avr).
Las observaciones críticas de Klement de que todas las cepas patógenas de Pseudomonas pueden desencadenar una rápida «respuesta de muerte celular hipersensible» (HR) en el sitio de inoculación de las hojas de tabaco, lo llevaron a sugerir que procesos moleculares similares regulan la virulencia y la resistencia. La tecnología del DNA recombinante desarrollada en los años 1970 y 1980 revolucionó este campo.
Un avance crucial fue el desarrollo de vectores de clonación de una amplia gama de huéspedes para investigar la biología de Rhizobium y la comprensión de que también podrían usarse para estudiar genes de Pseudomonas y Agrobacterium.
La idea conceptual clave fue que los genes Avr dominantes definidos genéticamente por Flor en un patógeno fúngico también podrían aislarse de bacterias patógenas identificando en una biblioteca de clones hechos de DNA de una raza patógena resistente, aquellos clones que confieren ganancia de HR inductora. fenotipos después de la administración en cepas de Pseudomonas virulentas (que no inducen HR).
El primer gen Avr se clonó a partir de una Pseudomonas que causaba enfermedades en algunos, pero no en todos, los cultivares de soja. Muchos otros siguieron.
La clonación de genes de avirulencia proporcionó importantes conocimientos sobre la biología efectora incluso antes de que se dilucidaran los mecanismos de su introducción en las células huésped. AvrBs2 de una cepa de Xanthomonas euvesicatoria patógena en pimiento fue el primer efector reconocido que se demostró necesario para una virulencia y aptitud total.
Esto sugirió que el gen R del pimiento correspondiente, Bs2, probablemente sería «duradero» porque la pérdida de la función AvrBs2 compromete la aptitud del patógeno. Es importante destacar que los aminoácidos AvrBs2 necesarios para el reconocimiento también lo son para la virulencia.
De hecho, el pimiento Bs2 confiere una fuerte resistencia al tomate transgénico y protege contra Xanthomonas, un patógeno grave en ambientes cálidos y húmedos.
Estos y otros hallazgos similares resolvieron la paradoja de por qué los patógenos codifican genes que tienen un gran costo de aptitud física en algunas plantas hospedantes; Estos genes codifican funciones de virulencia en otros genotipos del mismo huésped o de otras especies de huésped que no pueden reconocerlos.
Posteriormente, varios artículos demostraron que las proteínas Avr se reconocen dentro de la célula huésped. Esto sugirió que debe haber un mecanismo de administración y que las proteínas codificadas por el gen R probablemente sean intracelulares.
Finalmente, el hecho de que genes no relacionados de un patógeno de una planta huésped puedan ser reconocidos como genes Avr por una segunda especie de planta huésped proporcionó la primera pista de que las interacciones «gen por gen» probablemente contribuyeran a la restricción del rango de huéspedes y » resistencia no huésped”.
Paralelamente, la mutagénesis de transposones reveló mutantes de Pseudomonas que perdieron la capacidad de provocar HR en el tabaco.
De acuerdo con la hipótesis de Klement, estos mutantes también perdieron virulencia en su planta huésped nominal debido a que no pudieron entregar los efectores necesarios para causar la enfermedad.
Se hicieron descubrimientos simultáneos en patógenos bacterianos de animales y la secuenciación de estos grupos definió los sistemas de secreción de tipo III que están ampliamente distribuidos entre patógenos gramnegativos de plantas y animales.
Los sustratos de secreción de tipo III, incluidas las proteínas Avr, pasaron a denominarse «efectores». Posteriormente se demostró que muchos efectores bacterianos codifican enzimas que modifican dianas proteicas del huésped que contribuyen a las respuestas inmunitarias.
El término «efector» se adoptó para referirse a cualquier proteína patógena entregada al apoplasto o al interior de la célula vegetal.
La disección de la función efectora y la evolución, y la consiguiente definición de objetivos celulares del huésped, ha iluminado el campo durante los últimos 30 años.
Cuando los investigadores convergieron en la planta modelo Arabidopsis thaliana a mediados de la década de 1980, el progreso en la investigación del MPMI se aceleró (Figura 3).
Se identificaron dos cepas de Pseudomonas syringae (DC3000, un patógeno del tomate, y ES4326, un patógeno de Brassicas) que causan enfermedades en el genotipo Columbia de Arabidopsis de referencia (Col-0). Una vez que se identificaron los genes Avr clonados (ver arriba), se pudieron conjugar en DC3000 o ES4326 y probar las cepas resultantes para determinar la adquisición de reconocibilidad (avirulencia) en diversos genotipos de Arabidopsis.
Una vez que se identificaron las muestras con reconocimiento diferencial, ya sea en variación natural o a partir de poblaciones de plantas mutagenizadas, se pudo iniciar la genética vegetal para identificar el gen del receptor inmunológico correspondiente.
Además, a pesar de las primeras afirmaciones de que “Arabidopsis no tiene patógenos”, se identificaron y desarrollaron patosistemas naturales, en particular el patógeno del mildiú velloso de Arabidopsis, Hyaloperonospora arabidopsidis (Hpa; anteriormente Peronspora parasitica, un patógeno filamentoso oomiceto similar a un hongo relacionado con el patógeno del tizón tardío de la papa). Phytophthora infestans), Albugo spp (Ac) que causan roya blanca en Arabidopsis y Brassicas relacionadas, y mildiú polvoriento.
Estos descubrimientos permitieron el siguiente gran avance en MPMI, la identificación de genes R de resistencia a enfermedades que codifican receptores inmunes innatos intracelulares.
Reveladores de receptores inmunes, 1994-2004
El enfoque en Arabidopsis, con sus recursos en rápida expansión para el análisis del genoma y la clonación basada en mapas, permitió la identificación de múltiples receptores inmunes que detectan efectores de Pseudomonas (por ejemplo, RPS2, RPM1, RPS5 y el par RPS4-RRS1).
Se aprovechó la amplia variación natural entre las accesiones de Arabidopsis en su respuesta a razas específicas de Hpa para facilitar el aislamiento de un grupo de genes de resistencia a RPP durante este período. Todas las proteínas NLR codificadas (detalladas a continuación).
Si bien el tabaco, el tomate y el lino eran menos susceptibles que Arabidopsis a la clonación basada en mapas, el primer gen R clonado fue el Pto del tomate. Pto es una proteína quinasa y reconoce el efector de Pseudomonas AvrPto.
El gen N del tabaco que reconoce el virus del mosaico del tabaco, el Cf-9 del tomate que confiere resistencia al hongo Cladosporium fulvum y el alelo L6 del gen de resistencia a la roya del lino que reconoce razas específicas de la roya del lino se clonaron transformando plantas que portaban estos genes con el transposón Activador del maíz. o su derivada no autónoma Disociación.
Las poblaciones de plantas resultantes se examinaron en busca de mutantes que perdieran la función del receptor inmunológico y se identificó y analizó el locus de inserción del transposón, lo que permitió el aislamiento del gen R correspondiente.
La comparación de las secuencias de proteínas codificadas por los genes R descritos anteriormente permitió la definición de características conservadas de las proteínas NLR que hoy se reconocen como características distintivas de esta superfamilia.
Todos menos Pto portaban repeticiones ricas en leucina (LRR). Cf-9 y Xa21 son receptores de superficie celular con LRR extracelular es con (en arroz Xa21 para resistencia a Xanthomonas) o sin (Cf-9) un dominio de proteína quinasa intracelular.
Los otros codificaban proteínas modulares con un dominio de unión a nucleótidos (NB) y LRR C-terminales que en sus extremos N llevan un dominio de proteína Toll/Interleucina-1/Resistencia (TIR) (para N, L6, RPP1, RPP5, RPS4, y RRS1) o un dominio en espiral (CC) (para RPS2, RPM1, RPS5 y RPP13).
Los receptores inmunes intracelulares se parecen a los receptores de mamíferos como NOD1, NOD2 y NLRC4, y esta clase de proteína se denominó NLR (ya sea por receptores similares a NOD o por receptores repetidos ricos en leucina que se unen a nucleótidos).
Estructuras modulares similares son evidentes en proteínas apoptóticas (APAF1 y CED4), en proteínas de compatibilidad somática de hongos y en proteínas implicadas en la resistencia bacteriana a los bacteriófagos. Los mecanismos inmunitarios iniciados tras la detección de patógenos por receptores intracelulares llegaron a conocerse como «inmunidad desencadenada por efectores» (ETI).
Estas observaciones sientan las bases para nuestra comprensión actual de la inmunidad de las plantas basada en procesos iniciados tras la detección de ligandos patógenos por receptores de la superficie celular, receptores intracelulares o ambos.
Receptores de superficie e inmunidad activada por patrones (PTI)
Muchos patrones moleculares microbianos o asociados a patógenos (MAMPS o PAMPS) derivados de patógenos activan la inmunidad desencadenada por patrones (PTI) a través de los receptores de reconocimiento de patrones (PRR) de la superficie celular, como en la inmunidad innata de los animales, a menudo mediante el reconocimiento de los mismos P/MAMP.
La flagelina bacteriana, Pep13 de P. megasperma, el polímero fúngico quitina (principalmente sus productos de hidrólisis parcial en oligómeros de N-acetil glucosamina), oligogalacturónidos de paredes celulares vegetales (un patrón molecular asociado a daños, o DAMP) y muchos otros ligandos desencadenan reacciones similares. respuestas en las células que los perciben.
Estos incluyen la inducción de conjuntos de genes similares en respuesta a cada ligando, la producción de ROS a través de NADPH oxidasas y la activación de múltiples proteínas quinasas, incluidos al menos dos conjuntos de proteínas quinasas activadas por mitógenos o MAPK.
A pesar de los esfuerzos bioquímicos decididos en varios laboratorios, no se identificó ningún receptor PAMP/MAMP hasta que el laboratorio de Boller utilizó la genética para identificar el gen de detección de flagelina 2 (FLS2) de Arabidopsis.
FLS2 es el locus genético que codifica la capacidad de responder a la flagelina bacteriana o su sustituto truncado, flg22. FLS2 codifica un receptor de proteína quinasa LRR en la superficie de las células vegetales. La función FLS2 requiere el correceptor BAK1, otra proteína quinasa LRR definida originalmente como necesaria para la función de BRI1, una quinasa LRR que detecta y permite respuestas a la hormona vegetal brasinolida.
La proteína similar al receptor Cf-9 del tomate, tras el reconocimiento de la proteína apoplástica Avr9 de C. fulvum, un péptido del nudo de cistina secretado estrechamente plegado de 28 aminoácidos con 3 puentes disulfuro, también activa un conjunto de respuestas similares a PTI y requiere BAK1 para función. Avr9 fue la primera proteína de avirulencia fúngica definida. Cf-9 permite la percepción de Avr9 en tabaco y N. benthamiana, lo que facilita una investigación detallada mediante perfiles de expresión para revelar genes inducidos.
En particular, estos análisis revelaron ACIK1, el primero de muchos miembros de la familia de quinasas citoplasmáticas similares a receptores (RLCK) implicados en la inmunidad mediada por receptores de superficie. Arabidopsis BIK1 es el RLCK más estudiado y la definición de sus objetivos continúa revelando nuevos componentes de la maquinaria de defensa de la planta.
La activación de las NADPH oxidasas que generan ROS requiere la acción concertada de las RLCK y las proteínas quinasas dependientes de calcio; estas últimas probablemente se activan mediante el influjo de Ca2+ después de la activación del canal de calcio.
Entre muchos otros objetivos, los RLCK activan los canales de calcio que desempeñan funciones cruciales en la defensa de las plantas.
La señalización de PTI tiene bucles de amplificación mediados por varias fitocitocinas, que son reconocidas por proteínas similares a PRR que detectan péptidos derivados de plantas que se inducen o liberan al iniciar PTI. Otras LRR quinasas, RLCK, proteínas quinasas dependientes de calcio y proteínas fosfatasas sirven para atenúa o modula la PTI, lo que permite un ajuste exquisito de su fuerza y duración.
Se han definido muchos receptores extracelulares adicionales que reconocen ligandos bacterianos, fúngicos u otros ligandos.
La mayoría expresa ectodominios LRR, aunque se encuentran varios otros ectodominios en los PRR. Una paradoja duradera es que los homólogos de las quinasas del receptor LysM que activan la defensa en respuesta a la quitina fúngica también pueden activar programas de desarrollo simbiótico tras la detección de oligómeros o derivados que contienen N-acetilglucosamina.
Guardias, vigilantes y señuelos: cómo maximizar un repertorio de receptores limitado
El aislamiento de Pto proporcionó un enigma: ¿cómo proporciona una proteína quinasa capacidad de percepción? Esto se resolvió en un artículo histórico de Cell que demostró que la función de Pto (y la función del parálogo de Pto vinculado, Fen, que confiere sensibilidad al insecticida fentión) requiere un receptor inmune NLR intracelular, Prf. Así, en este caso, la evolución había favorecido la duplicación y divergencia de una proteína monitorizada por un NLR en lugar de la evolución de un NLR para percibir ligandos independientes que actúan a través de Pto o Fen.
Esto impulsó la propuesta de la «hipótesis de la guardia» en la que algunos NLR, en lugar de reconocer directamente los efectores patógenos, reconocen la modificación de una proteína del huésped por un efector patógeno.
Reconocimiento indirecto mediado por receptores de una molécula propia modificada que tiene un importante
La función de defensa y, por lo tanto, puede ser atacada repetidamente por efectores de virulencia de patógenos, puede ampliar un repertorio limitado de receptores inmunes codificados en la línea germinal, reduciendo la necesidad de desarrollar muchas especificidades de reconocimiento independientes únicamente a través de un repertorio NLR.
Muchos NLR funcionan protegiendo los componentes del host. Arabidopsis RPS2 y RPM1 monitorean el estado de la proteína RIN4, a la que se dirigen siete efectores bacterianos de tipo III no relacionados. RPS2 activa la inmunidad tras la escisión de RIN4 por la proteasa AvrRpt2 y RPM1 activa la inmunidad al detectar modificaciones postraduccionales mediadas por AvrB o AvrRpm1 de RIN4 que, en última instancia, perturban el estado del interruptor fosfórico de RIN4. RPS5 monitorea el estado de RLCK PBS1; cuando la proteasa AvrPphB escinde PBS1, RPS5 activa la inmunidad. ZAR1 monitorea el estado de varias proteínas similares a RLCK a través de pseudoquinasas intermedias como ZED1 o RKS1. ZAR1 se destaca por ser uno de los pocos NLR que muestra homólogos en especies de plantas emparentadas lejanamente.
Es importante destacar que es probable que algunos NLR de mamíferos también respondan a la acción de efectores de patógenos.
Si un componente protegido sufre una mutación, el resultado puede ser autoinmunidad debido a la desrepresión del NLR.
No todos los genes R genéticamente definidos codifican proteínas NLR. Una clase novedosa e intrigante de genes R codifica proteínas de fusión de proteína quinasa, incluidas proteínas quinasas en tándem o proteínas quinasas fusionadas a otros dominios.
En el trigo y la cebada, varios genes R codifican genes de quinasa en tándem que comprenden una quinasa funcional y una pseudoquinasa. Posiblemente, estas quinasas polimórficas podrían estar protegidas por un NLR relativamente conservado, análogo a Pto-Prf o RKS1-ZAR1 detallados anteriormente.
En la superficie celular también se encuentran receptores que protegen los objetivos del huésped. Por ejemplo, el receptor de superficie celular de tomate Cf-2 que, al igual que Cf-9, confiere resistencia a C. fulvum, requiere una cisteína proteasa secretada, Rcr3, para conferir resistencia a las enfermedades. Rcr3 se une al efector apoplásico Avr2 de C. fulvum y es inhibido por él (que también inhibe otras cisteína proteasas secretadas por el huésped). Por lo tanto, Cf-2 protege a Rcr3 y activa la defensa cuando Avr2 lo inhibe.
Las cisteína proteasas de la familia Rcr3 contribuyen a la resistencia a las enfermedades, proporcionando una justificación evolutiva para su ataque por parte de inhibidores codificados por patógenos.
¿Todas las proteínas monitoreadas son componentes de defensa? El modelo señuelo señala que los huéspedes pueden desarrollar alelos o parálogos que ya no proporcionan funciones de defensa indispensables, sino que actúan como imitadores de auténticos objetivos patógenos.
Guardias, vigilantes y señuelos: cómo maximizar un repertorio de receptores limitado
El aislamiento de Pto proporcionó un enigma: ¿cómo proporciona una proteína quinasa capacidad de percepción? Esto se resolvió en un artículo histórico que demostró que la función de Pto (y la función del parálogo de Pto vinculado, Fen, que confiere sensibilidad al insecticida fentión) requiere un receptor inmune NLR intracelular, Prf. Así, en este caso, la evolución había favorecido la duplicación y divergencia de una proteína monitorizada por un NLR en lugar de la evolución de un NLR para percibir ligandos independientes que actúan a través de Pto o Fen.
Esto impulsó la propuesta de la «hipótesis de la guardia» en la que algunos NLR, en lugar de reconocer directamente los efectores patógenos, reconocen la modificación de una proteína del huésped por un efector patógeno.
Reconocimiento indirecto mediado por receptores de una molécula propia modificada que tiene un importante La función de defensa y, por lo tanto, puede ser atacada repetidamente por efectores de virulencia de patógenos, puede ampliar un repertorio limitado de receptores inmunes codificados en la línea germinal, reduciendo la necesidad de desarrollar muchas especificidades de reconocimiento independientes únicamente a través de un repertorio NLR.
Muchos NLR funcionan protegiendo los componentes del host. Arabidopsis RPS2 y RPM1 monitorean el estado de la proteína RIN4, a la que se dirigen siete efectores bacterianos de tipo III no relacionados. RPS2 activa la inmunidad tras la escisión de RIN4 por la proteasa AvrRpt2 y RPM1 activa la inmunidad al detectar modificaciones postraduccionales mediadas por AvrB o AvrRpm1 de RIN4 que, en última instancia, perturban el estado del interruptor fosfórico de RIN4. RPS5 monitorea el estado de RLCK PBS1; cuando la proteasa AvrPphB escinde PBS1, RPS5 activa la inmunidad. ZAR1 monitorea el estado de varias proteínas similares a RLCK a través de pseudoquinasas intermedias como ZED1 o RKS1. ZAR1 se destaca por ser uno de los pocos NLR que muestra homólogos en especies de plantas emparentadas lejanamente.
Es importante destacar que es probable que algunos NLR de mamíferos también respondan a la acción de efectores de patógenos. Si un componente protegido sufre una mutación, el resultado puede ser autoinmunidad debido a la desrepresión del NLR.
No todos los genes R genéticamente definidos codifican proteínas NLR. Una clase novedosa e intrigante de genes R codifica proteínas de fusión de proteína quinasa, incluidas proteínas quinasas en tándem o proteínas quinasas fusionadas a otros dominios.
En el trigo y la cebada, varios genes R codifican genes de quinasa en tándem que comprenden una quinasa funcional y una pseudoquinasa. Posiblemente, estas quinasas polimórficas podrían estar protegidas por un NLR relativamente conservado, análogo a Pto-Prf o RKS1-ZAR1 detallados anteriormente.
En la superficie celular también se encuentran receptores que protegen los objetivos del huésped. Por ejemplo, el receptor de superficie celular de tomate Cf-2 que, al igual que Cf-9, confiere resistencia a C. fulvum, requiere una cisteína proteasa secretada, Rcr3, para conferir resistencia a las enfermedades. Rcr3 se une al efector apoplásico Avr2 de C. fulvum y es inhibido por él (que también inhibe otras cisteína proteasas secretadas por el huésped). Por lo tanto, Cf-2 protege a Rcr3 y activa la defensa cuando Avr2 lo inhibe.
Las cisteína proteasas de la familia Rcr3 contribuyen a la resistencia a las enfermedades, proporcionando una justificación evolutiva para su ataque por parte de inhibidores codificados por patógenos.
¿Todas las proteínas monitoreadas son componentes de defensa? El modelo señuelo señala que los huéspedes pueden desarrollar alelos o parálogos que ya no proporcionan funciones de defensa indispensables, sino que actúan como imitadores de auténticos objetivos patógenos.
Biología estructural de los receptores inmunes: se revelan los resistosomas
En los últimos cinco años se han visto avances espectaculares en la comprensión de los mecanismos de las proteínas NLR, gracias a las mejoras en la biología estructural permitidas por los métodos crioEM (Figura 4). En la mayoría de los ejemplos, el dominio NB de las proteínas NLR se une al ADP en estado inactivo y, tras el reconocimiento de un efector patógeno, un cambio conformacional permite el intercambio de ADP por ATP.
Cuando se unen a ATP, los dominios NB pueden asociarse entre sí, lo que impulsa la formación de oligómeros que imponen proximidad inducida en el dominio de señalización N-terminal e inician procesos de señalización.
¿Qué pasa después? Componentes de señalización entre percepción y defensa.
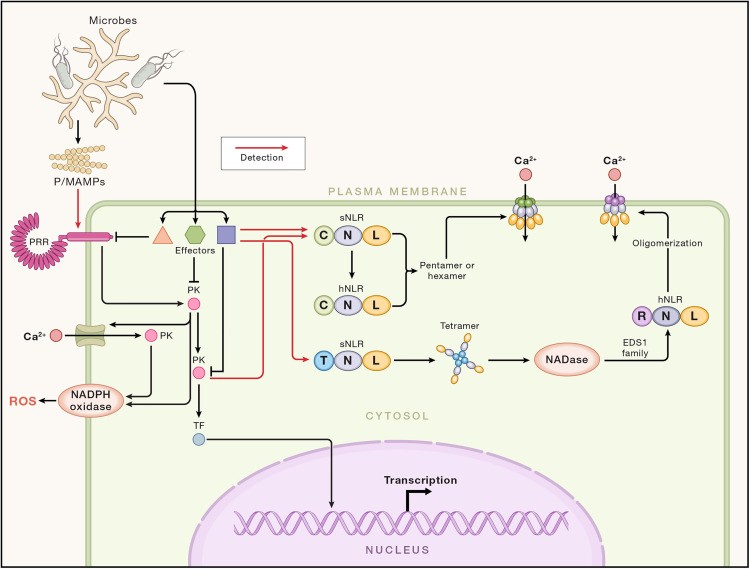
El análisis genético reveló componentes importantes para la señalización de defensa (Figura 5). En Arabidopsis se requieren genes de susceptibilidad mejorada a enfermedades 1 (EDS1) y de resistencia a enfermedades no específicas 1 (NDR1), respectivamente, para el funcionamiento completo de los genes TNL y CNL R.
También se identificaron mutantes que hacían que las plantas susceptibles fueran aún más susceptibles. Estas pruebas definieron la deficiencia de fitoalexina 4 (PAD4) y los genes implicados en la biosíntesis y la capacidad de respuesta a la hormona de defensa ácido salicílico (SA). NPR1, necesaria para la percepción de SA, y la isocorismato sintasa (ICS1), necesaria para la biosíntesis de SA, surgieron de dicha pantalla (ver más abajo).
Según los estudios de interacción de proteínas, EDS1 interactúa con PAD4 o con una tercera proteína de estructura similar, SAG101. El análisis comparativo de EDS1, PAD4 y SAG101 muestra que comparten un dominio similar a la lipasa (aunque sin actividad lipasa aparente) y un dominio «EP» compartido adicional. Sus funciones se detallan a continuación.
Una búsqueda diferente de componentes de señalización NLR implicó un estudio pionero de genética inversa utilizando silenciamiento genético inducido por virus (VIGS). Se construyó una biblioteca de DNAc de tabaco en un vector viral y luego se analizó en busca de clones que, cuando se silenciaban, comprometían la función del TNL N del tabaco que confiere resistencia al virus del mosaico del tabaco (TMV). Sorprendentemente, esto reveló el gen del requisito N (NRG1) que codifica una proteína CNL necesaria para que funcione una TNL.
Su dominio CC N-terminal muestra una homología pronunciada con RPW8, una proteína enigmática identificada como necesaria para la resistencia al mildiú polvoriento. NRG1 ahora se considera un miembro pionero de una clase de «NLR auxiliares», NLR cuya función es apoyar la función de “sensores NLR” involucrados en la detección de patógenos. NRG1 está estrechamente relacionado con la clase ADR1 de NLR auxiliares que también lleva un terminal N similar a RPW8; estas proteínas se conocen como RNL.
En Arabidopsis y otras dicotiledóneas, EDS1 y PAD4 funcionan con los NLR auxiliares de clase ADR1 para activar la defensa dependiente de la transcripción y EDS1 y SAG101 funcionan con la clase NRG1 de NLR auxiliares para promover la muerte celular durante la defensa.
Hay dos parálogos de NRG1 y tres parálogos de ADR1 en Arabidopsis Col-0, lo que explica por qué no se identificaron en las pruebas genéticas directas. Las funciones NRG1 y ADR1 son desigualmente redundantes.
Los RNL descritos hasta la fecha no son activados por moléculas codificadas por patógenos. Más bien, se activan mediante la activación de los sensores NLR aguas arriba.
La actividad NADasa de los tetrámeros de TNL genera pequeñas moléculas derivadas de nucleótidos que actúan sobre los heterodímeros de EDS1 con PAD4 o SAG101 y estimulan su interacción con ADR1 o NRG1 respectivamente, antes de eventos de señalización posteriores.
El trabajo actual se centra en EDS1-PAD4- Heterotrímeros ADR1 y EDS1-SAG101-NRG1 y sus estructuras. Posiblemente, la rotación del dominio EP PAD4 o SAG101 hacia afuera del cuerpo del heterodímero tras la unión del ligando crea una nueva interfaz para la interacción con ADR1 o NRG1, respectivamente.
Quedan preguntas importantes sobre los eventos posteriores que en última instancia conducen a la formación de oligómeros ADR1 o NRG1, potencialmente en distintas ubicaciones subcelulares con distintas funciones.
Una pregunta abierta crítica es cómo se produce la especialización de EDS1-PAD4-ADR1 y EDS1-SAG101. -Los módulos de señalización NRG1 promueven preferentemente la producción transcripcional y la muerte celular, respectivamente.
Se sabe desde hace mucho tiempo que la entrada de calcio en la célula es un marcador muy temprano de defensa y activación de NLR. Se identificaron muchos canales de Ca2+ como candidatos para este influjo temprano de calcio, pero no se demostró que ninguno fuera necesario.
Ahora es evidente que los NLR activados pueden funcionar como canales de Ca2+. Tanto las proteínas ZAR1 como las NRG1/ADR1 RNL pueden funcionar como canales autónomos de Ca2+ in vitro o in vivo, respectivamente. Las RNL de Arabidopsis actúan corriente abajo y son activadas por pequeñas moléculas derivadas de nucleótidos, dependientes de NADasa y generadas por TIR que a su vez activan los heterodímeros EDS1-PAD4 o EDS-SAG101 (ver arriba).
Por lo tanto, tanto la activación directa de las CNL como la activación indirecta de las RNL por moléculas pequeñas dependientes de TNL dan como resultado la actividad del canal de Ca2+.
Los datos combinados proporcionan una explicación mecanicista de cómo los receptores inmunes innatos TNL y CNL inician la muerte celular provocada por el reconocimiento de patógenos.
En Solanaceae, otra clase de NLR auxiliares, llamados NRC, soportan la función de casi la mitad de los sensores NLR. Los NRC se encuentran en todas las plantas de asteridos. Originalmente definidos como necesarios para la función de Cf-9, varios NRC admiten de forma redundante la función de conjuntos de sensores NLR ascendentes que se superponen de forma incompleta.
Por ejemplo, algunas funciones de sensor NLR requieren NRC2, NRC3 y NRC4, mientras que otras requieren NRC2 y NRC3, o simplemente NRC4.
Los extremos N de los NRC llevan un motivo N-terminal conservado y funcionalmente equivalente: el dominio MADA, que es funcionalmente intercambiable con el de ZAR1.
La activación de NRC tras la detección del ligando NLR del sensor provoca la formación de un oligómero NRC del cual se excluye el sensor NLR. Datos recientes sugieren que se forman los NRC de un dímero en el estado de preactivación y el oligómero activado (sorprendentemente un hexámero, no un pentámero). un canal de Ca2+ (Figura 4).
Pares de receptores inmunológicos y dominios de «señuelo integrado»
Algunos NLR funcionan en pares. El aislamiento de Arabidopsis RPP2 reveló el primer ejemplo de dos genes que codifican NLR, ambos necesarios para conferir capacidad de percepción, pero aún no está claro cómo actúa este par. Alrededor del 5 al 10% de los NLR en muchas especies están codificados en genes cabeza a cabeza y se cotranscriben.
Los TIR-NLR de Arabidopsis RPS4 y RRS1 comprenden un par bien caracterizado, definido respectivamente como necesario para el reconocimiento de Pseudomonas AvrRps4 y Ralstonia PopP2, una acetiltransferasa. Las accesiones que responden a PopP2 llevan el alelo RRS1-R y Col-0 lleva un alelo más corto. Alelo RRS1-S. RRS1-R también confiere resistencia a hongos del género Colletotrichum mediante el reconocimiento de un efector aún no caracterizado.
Los TIR-NLR de RRS1 contienen cerca de su extremo C un dominio de factor de transcripción WRKY integrado, y las proteínas del dominio WRKY están fuertemente implicadas en la defensa de las plantas. Sorprendentemente, se requiere RPS4 para que RRS1-R reconozca PopP2, y se requiere RRS1-S- o RRS1-R para que RPS4 reconozca AvrRps4.
Los genes RPS4 y RRS1 son adyacentes y se transcriben de forma divergente. Las proteínas RPS4 y RRS1 se asocian constitutivamente y forman un complejo de receptor inmunológico en el que el dominio WRKY de RRS1 detecta efectores de patógenos y la señalización de defensa se transduce a través de RPS4.
El dominio TIR de RRS1 es enzimáticamente inactivo. Se puede considerar que sensores como RRS1 y sus parálogos están protegidos por sus respectivos socios ejecutores, ejemplificados por RPS4 y sus parálogos.
Se han encontrado muchos ejemplos de esta disposición, y el dominio de detección del sensor se ha denominado dominio señuelo integrado (ID). En arroz, se definieron muchos NLR emparejados codificados adyacentemente, como RGA4 y RGA5, o Pik-1 y Pik-. 2 que confieren resistencia al patógeno del añublo del arroz Magnaporthe oryzae.
Tanto para RGA5 como para Pik-2, el ID es un dominio proteico asociado a metales pesados (HMA) que interactúa directamente con efectores de M. oryzae.
Curiosamente, un gen de resistencia recesivo, pi21, codifica una proteína de dominio HMA, una clase de proteína a la que se dirigen muchos efectores del añublo del arroz.
Se han seleccionado pares de receptores inmunes que contienen HMA ID que detectan la interacción de efectores con proteínas del dominio HMA. En un avance notable, el dominio HMA de Pik-2, que se encuentra entre el dominio CC y el NB, fue reemplazado por nanocuerpos que reconocen GFP o RFP y condicionan la resistencia a los virus etiquetados con GFP o RFP, acercando el sueño de poder diseñar receptores inmunes que puedan reconocer cualquier efector.
Diversidad de receptores inmunológicos
Las proteínas NLR protegen las moléculas de las plantas y responden cuando han sido modificadas por la acción de efectores de patógenos o reconocen directamente una proteína efectora.
Los receptores inmunitarios que interactúan directamente con los efectores de patógenos están codificados por genes que muestran una variabilidad genética mucho mayor que los receptores inmunitarios que protegen los componentes del huésped.
Los monocultivos de cultivos son propensos a epidemias de enfermedades. Aunque cualquier variedad de cultivo porta entre cientos y miles de genes que codifican receptores inmunes, si cada planta de una población es genéticamente idéntica, un patógeno aislado que pueda evadir la detección por el repertorio NLR de esa variedad puede colonizar toda la población de plantas.
Por el contrario, las poblaciones de plantas naturales genéticamente diversas son menos propensas a epidemias causadas por patógenos coevolucionados, aunque los patógenos importados pueden causar enfermedades en la mayoría de los huéspedes.
Los análisis de pangenomas verificaron que los repertorios de genes que codifican NLR muestran una amplia diversidad en comparación con otros genes. Hamilton et al. propusieron que la principal ventaja selectiva de retener la reproducción sexual y el cruzamiento es que el polimorfismo en los loci que contribuyen al reconocimiento del parásito restringe la enfermedad, y que «barajar la baraja» de la capacidad de reconocimiento de patógenos en cada generación crea nuevas combinaciones que un patógeno debe superar.
Según este modelo de selección dependiente de la frecuencia, si una población huésped es extremadamente heterogénea en su capacidad de reconocimiento, entonces la mayoría de los aislados del parásito no podrán crecer en la mayoría de los individuos huéspedes de la población.
La recombinación asegura que el repertorio de receptores inmunes en cada individuo de la progenie no sea idéntico al repertorio de ninguno de sus padres.
Es más probable que este polimorfismo se pierda en poblaciones que se autofertilizan a menos que se mantenga mediante selección. Además, si la recombinación sexual entre parásitos conduce al intercambio de genes efectores dominantes reconocidos, entonces la mayor parte de la progenie de la mayoría de los parásitos no podrá encontrar un huésped que no pueda reconocerlos.
La restricción del éxito de los parásitos en mezclas de variedades de plantas es consistente con este concepto general, y el enfoque merece una mayor exploración como estrategia para proporcionar una resistencia más duradera a las enfermedades. Este pensamiento también enfatiza el desafío de reclutar repertorios de receptores inmunes que confieren resistencia duradera en monocultivos.
La extraordinaria diversidad de factores de virulencia procariotas.
Una función común de los efectores secretados bacterianos de tipo III (T3E) es alterar las respuestas inmunitarias de las plantas iniciadas por los PRR de la superficie celular o los receptores NLR intracelulares. Muchos T3E son enzimas, imitadores de enzimas o inhibidores de enzimas y, a menudo, se dirigen a módulos de señalización eucarióticos comunes.
La mutación de cualquier efector por lo general no logra reducir la virulencia en una planta susceptible. Muchos efectores de la enzima T3E no pueden perder la función que les permite ser reconocidos, lo que resulta en polimorfismos frecuentes de presencia/ausencia impulsados por la variación en los receptores inmunes del huésped.
El reconocimiento de las funciones de virulencia de T3E suele ser indirecto, como se detalló anteriormente. Debido a que el reconocimiento indirecto impulsa la selección para la pérdida del T3E infractor, los patógenos bacterianos a menudo desarrollan funciones independientes para atacar la misma proteína del huésped.
Por ejemplo, al menos siete efectores evolucionados independientemente se dirigen a la proteína RIN4 de la membrana plasmática intrínsecamente desordenada para alterar su función en el tráfico de vesículas.
Estos efectores modifican RIN4 ya sea mediante (i) proteólisis, que activa el NLR de RPS2 o (ii) ribosilación de ADP, que altera un sitio de fosforilación del huésped cercano y activa el NLR de RPM1 o (iii) mediante acetilación de componentes de la proteína RIN4-RPM1. complejo. Otras proteínas de defensa del huésped comúnmente atacadas incluyen los dominios quinasa de PRR importantes, como el receptor de flagelina FLS2 y su correceptor BAK1, y RLCK.
Los sistemas de reconocimiento indirecto están sujetos a cambios recurrentes por selección contra la acción efectora, pérdida de efectividad del NLR, evolución de un nuevo efector dirigido al mismo objetivo de defensa del huésped, evolución y reclutamiento de un nuevo NLR que reconoce la nueva modificación del objetivo, etc.
Las cepas de Pseudomonas syringae portan un pangenoma de alrededor de 70 familias efectoras. Estas suites brindan redundancia y cierta variación intrafamiliar que garantiza flexibilidad evolutiva.
El rango de huéspedes suele estar determinado por las consecuencias paralelas de los eventos de reconocimiento mediados por NLR.
Por lo tanto, cambiar de host podría requerir una pérdida casi simultánea de múltiples efectores, un escenario poco probable. Sólo HopM, AvrE, HopB y HopAA están codificados en >95% de las cepas, lo que sugiere su importancia fundamental.
De hecho, tres de estos cuatro efectores están codificados físicamente en un locus vinculado al propio sistema de secreción de tipo III.
De manera similar, el reconocimiento por parte de los huéspedes de efectores únicos, al menos en Pseudomonas syringae, puede tener efectos débiles y muchos efectores convergen en genes NLR únicos, probablemente a través de la focalización compartida de una proteína del huésped protegida. Por tanto, la virulencia es la suma de las funciones de ese conjunto de efectores totales entregados que encuentran un objetivo huésped en las células de una especie de planta determinada, e incluso un reconocimiento débil puede ser eficaz.
Los efectores se pueden organizar conceptualmente en «gremios» que se dirigen a procesos celulares del huésped relacionados. Existe una presión evolutiva para mantener un conjunto de efectores que cubra cada gremio necesario para superar la respuesta de defensa del anfitrión.
Una función crítica es proporcionar un ambiente apoplástico de alta humedad para apoyar el crecimiento bacteriano. HopM1 y AvrE tienen funciones redundantes vitales para este fenotipo. HopM1 ayuda a establecer un entorno extracelular acuoso probablemente manipulando el tráfico de vesículas del huésped mediado por la proteína MIN7, un objetivo de HopM1.
Los miembros de la familia AvrE codifican canales de agua o soluto y los inhibidores de moléculas pequeñas de AvrE bloquean la virulencia en un mutante HopM1.
Las Xanthomonas fitopatógenas (y también muchas cepas de Ralstonia) utilizan secreción de tipo III para administrar efectores TAL (similares a activadores de la transcripción) que se unen al ADN del huésped y activan genes de susceptibilidad específicos del huésped. Por ejemplo, PthXo1, un efector TAL del patógeno del arroz Xanthomonas oryzae, activa la expresión de un transportador de azúcar localizado en la membrana del arroz, la proteína DULCE Os8N3.
La susceptibilidad proporcionada por la expresión inapropiada de Os8N3 dependiente de TAL ha seleccionado variantes de arroz que portan mutaciones promotoras recesivas que previenen la unión de PthXo1. Se han utilizado métodos de edición de genes para alterar el promotor de Os8N3 y otros genes SWEET que son activados por otros efectores TAL, de modo que estos genes ya no se induzcan tras la infección y la resistencia sea elevada.
El descubrimiento de los efectores TAL también permitió diseñar un nuevo tipo de proteína de unión al ADN programable. La unión al ADN por cada efector TAL se especifica mediante secuencias en las repeticiones variables de 34 aminoácidos en cada proteína TAL.
El mayor polimorfismo se produce en las posiciones 12 y 13 de cada repetición, el llamado di-residuo de repetición variable (RVD). Los residuos de aminoácidos que representan los RVD de cada efector TAL corresponden a los nucleótidos en el sitio diana en el DNA de la planta.
Los efectores TAL pueden diseñarse para unirse a cualquier secuencia de DNA y pueden unirse a nucleasas u otros dominios.
Otros procariotas causan enfermedades importantes. Los efectores de virulencia de los fitoplasmas relacionados con micoplasmas revelaron nuevos mecanismos que promueven el éxito de los patógenos y atenúan la señalización de defensa al interferir con la abundancia o función del factor de transcripción del huésped.
Patógenos microbianos eucariotas: la genómica se encuentra con la biología estructural y Alphafold
Los microbios eucariotas filamentosos, incluidos los hongos ascomicetos y basidiomicetos y los oomicetos filogenéticamente distintos de Stramenopile, causan algunas de las enfermedades de cultivos más devastadoras.
Comparten las propiedades de producción prolífica de esporas y dispersión por el viento, germinación en una hoja seguida de colonización y rápida conversión de nutrientes del huésped en rondas posteriores de esporulación y colonización.
Muchos son biotrofos obligados que requieren un huésped vivo para prosperar, reproducirse y expresar cientos de genes efectores de virulencia candidatos. Algunos son hemibiotrofos, comienzan siendo biotróficos y luego cambian a un estilo de vida necrotrófico.
Muchos biotrofos y hemibiotrofos forman haustorios, una invaginación de la membrana plasmática del huésped que permite el contacto íntimo para la importación de nutrientes y la entrega de efectores entre el parásito y el huésped. Otros son puramente necrotróficos; estos despliegan toxinas para matar las células vegetales y alimentarse de su contenido.
A pesar de la importancia y el interés de los mecanismos de virulencia dependientes de toxinas, algunos de los cuales activan los NLR para promover la muerte de la célula huésped, las restricciones de espacio impiden una explicación extensa de la virulencia dependiente de toxinas.
En cambio, presentamos algunas enfermedades importantes y sus agentes causales y comentamos sobre la evolución de los efectores eucarióticos y su llegada a las células huésped.
Para todas estas enfermedades, la adquisición de secuencias completas de pangenomas, expresión genética y datos de polimorfismos durante los últimos 15 años ha sido fundamental para el descubrimiento.
Los hongos de la roya basidiomicetos colonizan la mayoría de las plantas (aunque no Arabidopsis). Los estudios pioneros de Flor se llevaron a cabo sobre la roya del lino y este sistema reveló una gran cantidad de conocimientos, lo que permitió el aislamiento de muchos alelos de cuatro loci de receptores NLR intracelulares del lino y efectores reconocidos de la roya.
La roya de la soja requiere la aplicación de 2.000 millones de dólares en fungicidas al año; Se están realizando esfuerzos vigorosos para identificar y desplegar nuevas fuentes genéticas de resistencia.
El trigo se ve afectado por tres royas del género Puccinia; P. graminis (roya del tallo), P. striiformis (roya amarilla o rayada) y P. triticina (roya de la hoja) y, en muchos lugares, las variedades de trigo están expuestas a las tres.
Se han identificado muchos genes R contra el mildiú polvoriento y la roya del trigo; la mayoría codifica proteínas NLR y a menudo exhibe series alélicas extensas e interesantes, como la cebada Mla, mientras que otras codifican las quinasas en tándem presentadas anteriormente, cuyo mecanismo de señalización inmune sigue siendo desconocido.
El patógeno oomiceto más notorio es Phytophthora infestans, el agente causal del tizón tardío de la papa y el tomate. Existen muchas otras especies de Phytophthora que causan enfermedades graves en otras plantas. Muchos genes R que codifican NLR se clonaron contra P. infestans y contra su pariente, el patógeno del mildiú velloso de Arabidopsis, Hpa (ver arriba).
Otros oomicetos causan pérdidas importantes en otros cultivos, por ejemplo el mildiú de la espinaca, la lechuga y la vid. Tanto P. infestans como los efectores Hpa llevan un péptido señal para la exportación desde el patógeno y uno o ambos de un «motivo RxLR» y un «motivo DEER» en el efector secretado.
La genómica reveló cientos de tales efectores en estos genomas (Figura 6). Se definieron los objetivos del huésped de algunos de estos efectores, lo que proporcionó conocimientos novedosos sobre los mecanismos de defensa y verificó funciones importantes para la señalización de la proteína quinasa, la función del canal, la autofagia y el silenciamiento de genes.
Muchos llevan repeticiones en tándem de dominios WY que se combinan para interactuar con las proteínas del huésped. En un informe reciente notable, un efector de Phytophthora WY (Figura 6B) interactúa con las proteínas fosfatasas de una manera que resulta en la supresión de los mecanismos de silenciamiento del gen del huésped.
Las bibliotecas de tales efectores facilitan la búsqueda de aquellos reconocidos por las proteínas NLR. Los efectores del patógeno oomiceto de la roya blanca, Albugo, comparten un motivo «CCG» cerca del extremo N de la porción secretada de la proteína.
El mildiú polvoriento es un biotrofo haploide de ascomicetos que coloniza solo las células epidérmicas de las hojas de plantas monocotiledóneas y dicotiledóneas.
La coevolución del trigo y la cebada con sus respectivos mildiú polvoroso específicos del huésped llevó a los criadores a seleccionar muchos genes R semidominantes diferentes que a menudo codifican receptores inmunes NLR.
Por el contrario, el mildiú polvoriento del tomate dicotiledóneo, las cucurbitáceas y la Arabidopsis muestran menos especialización en el huésped y se han identificado pocos genes R del mildiú polvoriento en estos grupos taxonómicos.
Para el mildiú polvoriento, los primeros efectores reconocidos revelaron una clase de proteína secretada que a menudo lleva un péptido post señal con motivo CxY y probablemente evolucionó a partir de una proteína RNasa. Sorprendentemente, los efectores de mildiú CxY no relacionados con la secuencia reconocidos por diferentes alelos Mla NLR comparten un pliegue común (Figuras 6K y 6L).
De manera similar, múltiples efectores reconocidos del patógeno del añublo del arroz Magnaporthe oryzae, muchos de los cuales interactúan con las proteínas del dominio HMA del huésped, comparten el pliegue efector «MAX» (Figuras 6E y 6F).
Estos descubrimientos sugirieron que los motivos efectores conservados pueden correlacionarse con la entrega a la célula vegetal, una idea originalmente respaldada por la expresión de efectores de hongos de roya dentro de las células vegetales y por la inmunohistoquímica de un efector de roya dentro de las células vegetales. Los hongos filamentosos probablemente entregan efectores intracelulares a las células vegetales mediante endocitosis.
Hasta la fecha, el sistema de administración mejor estudiado es el utilizado por Magnaporthe oryzae, donde los efectores apoplásicos se secretan mediante secreción convencional dependiente de Golgi. Por el contrario, los efectores que en última instancia se dirigen al citoplasma del huésped se administran primero a vesículas en una estructura especializada en la superficie de hifas fúngicas invasivas en crecimiento llamada complejo biotrófico-interfacial (BIC) a través de una ruta independiente de Golgi.
El BIC se forma después de la penetración exitosa de la primera célula vegetal colonizada. Todos los efectores citoplasmáticos analizados hasta la fecha se localizan en el BIC.
La clatrina y actina vegetales se colocalizan con vesículas que contienen efectores fúngicos y la reducción de su función impide la liberación de efectores.
Los experimentos de silenciamiento génico e inhibidores dirigidos a clatrina vegetal y componentes de vesículas endocíticas de clatrina comprometen la generación de vesículas que contienen efectores y los consiguientes síntomas del añublo del arroz, mientras que el silenciamiento de la endocitosis independiente de clatrina no lo hizo.
Se demostraron resultados similares al estudiar la administración de efectores desde los haustorios que invaginan las células huésped del patógeno oomiceto Phytophthora infestans, lo que sugiere que estos procesos podrían conservarse. Por lo tanto, diferentes mecanismos entregan efectores al apoplasto o al citoplasma del huésped, y la endocitosis mediada por clatrina surge como un mecanismo probable para este último.
Sin embargo, se puede esperar una mayor diversidad en la administración de efectores fúngicos a las células vegetales, ya que los patógenos que causan enfermedades del carbón, como Ustilago maydis, parecen expresar un conjunto conservado de genes que probablemente codifican una estructura translocón para administrar efectores a la célula huésped.
Se han definido varios efectores reconocidos de los hongos de la roya. Sin embargo, aún no se han definido motivos de secuencia de diagnóstico para inspeccionar el secretoma de las razas secuenciadas. Además, los notables avances computacionales que permitieron Alphafold ahora se pueden aplicar a la investigación de familias de efectores basándose en la predicción estructural y esto está abriendo nuevas puertas para comprender los repertorios de efectores.
Finalmente, muchos invertebrados, como los nematodos y los pulgones, mantienen interacciones prolongadas con sus plantas hospedantes y, al igual que los microbios, liberan efectores que influyen en la biología del hospedador. Se seguirán trabajando para revelar nuevos conocimientos sobre los efectores de patógenos eucariotas y sus mecanismos de inhibición de defensa.
Integrando marcos conceptuales-potenciación mutua de PTI y ETI
PTI, la cadena de eventos que sigue a la detección del ligando por los PRR de la superficie celular, está bien documentada. Sin embargo, hasta hace poco no estaba disponible ningún análisis de eventos específicos de ETI porque los ensayos de ETI generalmente se realizaban en presencia de PTI. Los efectores normalmente se administraban a las células vegetales mediante sistemas de administración microbianos que inevitablemente también desencadenan la PTI.
La expresión inducible de efectores reconocidos en ausencia de PTI permite la investigación de ETI únicamente. Tras la activación del receptor inmunitario NLR mediante la inducción de un efector reconocido sin PTI, se eleva la abundancia de proteínas que contribuyen a la PTI y se coinducen rápidamente genes de respuesta inmunitaria y genes de biosíntesis de hormonas de defensa.
En ausencia de PTI, la activación de ETI produce menos muerte celular que en presencia de PTI. Esta potenciación mutua de PTI y ETI es indispensable para una activación de defensa sólida.
Además, el nodo EDS1-PAD4-ADR1 es un punto de convergencia para las cascadas de señalización de defensa activadas por receptores LRR intracelulares y residentes en la superficie y la señalización ETI aumenta las salidas de PTI.
Se requiere PTI para que el NLR NRG1 auxiliar se oligomerice después de la activación de TIR-NLR. Estos hallazgos son consistentes con el hecho de que la mayoría de los genes R que codifican NLR son semidominantes, lo que implica que la cantidad cuantitativa de activación de la defensa se controla a través de la abundancia del receptor inmunológico.
Una conclusión clave de este trabajo es que (1) son los mecanismos de PTI los que frustran el éxito de los patógenos; (2) ETI restaura y reinicia PTI después de su atenuación por efectores patógenos o durante la autorregulación; y (3) más receptores inmunes, conferidos por pilas de genes R, deberían permitir una activación de defensa más fuerte, además de hacer que sea más difícil para los patógenos evadir la detección mediante la eliminación de múltiples efectores reconocidos.
Los profundos orígenes evolutivos de la inmunidad a las angiospermas.
Las plantas colonizaron la tierra por primera vez hace aproximadamente 500 millones de años y los restos de plantas fosilizadas sugieren la presencia de estructuras haustoriales consistentes con la colonización fúngica (probablemente por hongos micorrízicos simbióticos) de estos primeros colonizadores.
El análisis del genoma de parientes de las primeras plantas terrestres, como musgos y hepáticas, reveló homólogos de muchos componentes del receptor inmunológico de angiospermas y los estudios funcionales muestran que los RLCK y los PRR del dominio LysM contribuyen a la resistencia a los patógenos. También se identificaron múltiples arquitecturas NLR N-terminales, incluido un dominio N-terminal alfa/beta hidrolasa generalizado que no se encuentra en las NLR de angiospermas.
En términos generales, la inmunidad mediada por receptores de superficie e intracelulares ha contribuido durante mucho tiempo a la resistencia a patógenos en las plantas terrestres.
Los tamaños del repertorio de receptores inmunes varían enormemente, con muy pocos en las plantas acuáticas, y los tamaños del repertorio de los receptores de superficie e intracelulares varían de manera coordinada. Los motivos cruciales para la interacción del correceptor en LRR-RLP están estrechamente relacionados con los del subgrupo Xb de la quinasa similar al receptor LRR (RLK), que percibe fitohormonas y gobierna principalmente el crecimiento y el desarrollo.
Estos datos sugieren que los receptores de la superficie celular implicados en la inmunidad y el desarrollo comparten un origen común.
¿Cuál es la base de la resistencia “no huésped”?
A pesar de la vulnerabilidad de los monocultivos a las enfermedades (ver antes), la especialización en patógenos significa que los monocultivos de una especie rara vez sucumben a patógenos que colonizan una especie estrechamente relacionada.
Así, los monocultivos de trigo son resistentes a razas de oídio o roya lineal que infectan a la cebada, y viceversa. ¿Cuál es la base de esta resistencia ajena al huésped? ¿Implica los mismos mecanismos que sustentan la variación en la resistencia a patógenos adaptados (PTI y ETI), o implica mecanismos adicionales y distintos?
En una revisión profética, Panstruga y Schulze-Lefert predijeron que la resistencia a través de procesos dependientes de NLR probablemente explicaría por qué las especies no hospedantes son resistentes a patógenos de especies estrechamente relacionadas, pero las diferencias en los receptores de superficie y otros mecanismos pueden explicar la resistencia a patógenos de especies estrechamente relacionadas. especies más lejanamente emparentadas.
En términos generales, esta predicción ha sido validada. Por ejemplo, no existen Xanthomonas patógenos del tabaco, pero Xanthomonas euvesicatoria puede crecer en N. benthamiana si el reconocimiento del efector XopQ por el afín Roq1 TNL NLR se pierde por mutación en el huésped o patógeno.
Las razas de Albugo candida que causan enfermedades en Brassicas pero no pueden infectar a Arabidopsis son resistidas por múltiples receptores inmunes TIR-NLR en Arabidopsis que fueron disecados mediante segregación transgresiva. La resistencia de la cebada a la roya lineal del trigo involucra múltiples loci, al menos uno de los cuales codifica un receptor inmunológico NLR.
Los receptores de la superficie celular también pueden restringir el rango de huéspedes; el EFR PRR de Arabidopsis que reconoce la proteína bacteriana EF-Tu puede conferir una resistencia elevada a Ralstonia en la especie de tomate solanácea lejanamente relacionada que carece de esta capacidad de reconocimiento.
Estrategias para una resistencia duradera
Sabiamente se dice “nunca apuestes contra el patógeno”. El número de poblaciones de patógenos supera con creces el de las plantas hospedantes, y es probable que la resistencia dependiente del reconocimiento contra patógenos que portan cientos de genes efectores con funciones redundantes se pueda evadir fácilmente con suficientes mutaciones y recombinaciones. Y, sin embargo, la mayoría de las plantas son resistentes a la mayoría de los patógenos; la susceptibilidad a enfermedades es la excepción.
¿Cuáles son las perspectivas de convertir las plantas de cultivo de huéspedes a no huéspedes de sus importantes enfermedades? Dado que muchos genes R son semidominantes y PTI y ETI se potencian mutuamente, el reconocimiento simultáneo de repertorios efectores de patógenos por parte de múltiples NLR debería permitir una resistencia cuantitativamente más fuerte que un solo NLR.
Por lo tanto, las pilas de NLR funcionalmente validadas que reconocen distintos efectores deberían conferir un fortalecimiento fisiológico de la activación de la defensa, así como un beneficio genético resultante del requisito de múltiples mutaciones en un patógeno para evadir la detección por múltiples receptores inmunes.
La combinación de un casete de cinco transgenes que confiere resistencia de amplio espectro a un patógeno fúngico de la roya en el trigo es muy prometedora, al igual que una combinación triple de genes NLR para la resistencia al tizón tardío en la papa.
Un enfoque distinto es plantar genotipos de cultivos mixtos que imiten la diversidad poblacional de las poblaciones naturales. Sin embargo, las variedades vegetales vendidas a los agricultores deben cumplir requisitos de distinción, uniformidad y estabilidad.
Posiblemente, en un fondo genético común, se podrían proporcionar múltiples genes NLR distintos y las variedades que portan mezclas de tales líneas ralentizarían la evolución de la virulencia que supera cualquier combinación de capacidades de reconocimiento distintas en una población.
Para salvaguardar una resistencia duradera a las enfermedades, es importante desarrollar tecnologías para definir todos los efectores en una población de campo de un patógeno.
Esta información es crucial para definir qué genes R incorporar en las pilas de genes. Además, la combinación de pilas de genes R con mutaciones en los genes de susceptibilidad a enfermedades (S) puede proporcionar una capa adicional de durabilidad.
Tales mutaciones en el huésped reducirían las poblaciones de patógenos y reducirían la probabilidad de que puedan ocurrir mutaciones espontáneas en genes efectores de patógenos que superarían a los genes R afines. Posiblemente se podría insertar la pila del gen R en un gen de susceptibilidad a enfermedades.
Esto podría proporcionar un locus único que confiera múltiples mecanismos de resistencia que podrían introducirse fácilmente en diferentes cultivares.
Señalización Sistémica
Ross demostró que las infecciones virales estaban restringidas por infecciones previas. Esta resistencia también fue ampliamente eficaz contra patógenos adicionales. Ross acuñó el término “resistencia sistémica adquirida” (SAR) para la resistencia sistémica inducible y “resistencia adquirida localizada” para la resistencia inducida en hojas inoculadas adyacentes al sitio de inoculación.
La SAR se correlaciona con la acumulación de ácido salicílico (SA) y se ve comprometida si se previene la acumulación de SA.
El tratamiento con SA induce un conjunto de genes que incluyen aquellos que codifican proteínas secretadas relacionadas con la patogénesis (PR), algunas de las cuales están implicadas de manera creíble en la restricción del crecimiento de patógenos.
En Arabidopsis, una prueba de detección de mutantes que no responden a SA reveló el gen NPR1 y sus diversos parálogos. En una serie de elegantes experimentos, el laboratorio Dong arrojó luz sobre cómo NPR1 se relaciona con SA y con factores de transcripción TGA para activar genes que responden a SA, sobre cómo los cambios redox liberan monómeros de NPR1 que pueden ingresar al núcleo después de la interrupción de un oligómero citoplasmático de NPR1 y cómo NPR1 en el citoplasma puede dirigir la degradación dependiente de la ubiquitinación de proteínas que regulan negativamente la defensa.
Una molécula de señalización de defensa adicional, el ácido N-hidroxipipecólico (NHP), a menudo sintetizada con un curso temporal similar al SA, se ha implicado en la resistencia a las enfermedades. Su receptor aún se desconoce y las formas en que se cruzan las señales de NHP y SA siguen siendo un tema de investigación activo. Con la elección correcta del promotor y la regulación traslacional para minimizar la expresión inapropiada, NPR1 también se puede utilizar para elevar la resistencia a enfermedades de los cultivos sin incurrir en una penalización en el rendimiento.
Las contribuciones revolucionarias de CRISPR para revelar y desplegar la resistencia a las enfermedades
La facilidad con la que se pueden construir y administrar nucleasas CRISPR programables de ARN ha acelerado la edición de genes vegetales. CRISPR permite a los investigadores eliminar genes con precisión para validar genes candidatos implicados en la resistencia a enfermedades.
Por ejemplo, los genes candidatos para los QTL principales pueden validarse mutándolos y puntuando su fenotipo. Las mutaciones cuya función se identificó en una especie de planta se pueden realizar en genes ortólogos en cualquier especie de planta.
El ejemplo más notable es el gen mlo en la cebada, donde la eliminación del gen ortólogo en el trigo y el tomate da como resultado plantas resistentes al mildiú polvoriento.
Es importante destacar que la edición de genes CRISPR permite realizar mutaciones útiles en plantas poliploides en la generación T0. Un ejemplo similar es el gen dmr6 que se descubrió en Arabidopsis thaliana y permitió la construcción de mutaciones ortólogas en tomate, plátano, arroz y cítricos.
Con el tiempo, se puede prever la inserción de pilas de genes cisgénicos en ubicaciones precisas del genoma mediante la reparación dirigida por homología (HDR).
La implementación de esta tecnología en cultivares de élite ya mejorados para mejorar el rendimiento eliminará la resistencia al ligamiento y reducirá el tiempo necesario para introducir nuevas características.
El progreso en el campo MPMI no muestra signos de desaceleración. En los próximos años se verá más biología estructural de los receptores inmunes y predicción basada en IA de interacciones proteína/proteína, combinada con genómica, para enriquecer dramáticamente nuestra comprensión de cómo las plantas resisten las enfermedades y cómo los patógenos eluden las respuestas inmunes del huésped.
Prevemos la ingeniería de proteínas de NLR para ampliar el reconocimiento de nuevas proteínas efectoras. ¡Nuestra predicción más segura es que habrá nuevos conocimientos sorprendentes y emocionantes!
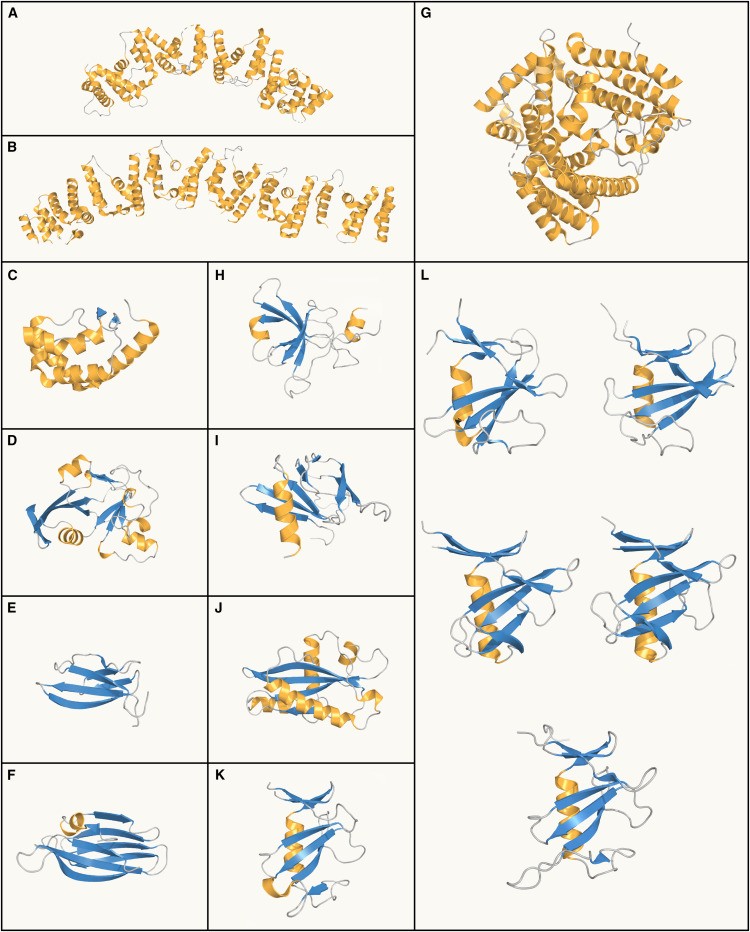
(A) Estructura determinada experimentalmente del efector RxLR PcRxLR12 de Phytophthora capsici (PDB: 5ZC3).
(B) Estructura determinada experimentalmente del efector PSR2 del dominio WY de Phytophthora sojae (PDB:5GNC).
(C) Estructura determinada experimentalmente de la beta-cinamomina elicitina de Phytophthora cinnamomi (PDB:1LJP).
(D) Estructura prevista del supuesto efector similar a ADP-ribosa transferasas de Magnaporthe oryzae (MGG_16989).
(E) Estructura determinada experimentalmente del efector MAX AVR1-CO39 de Magnaporthe oryzae (PDB:5ZNG).
(F) Estructura determinada experimentalmente de ToxA de Pyrenophora tritici-repentis (PDB:1ZLD).
(G) Estructura determinada experimentalmente de AvrSr35 de Puccinia graminis f. sp. tritici (PDB:7XC2).
(H) Estructura prevista del supuesto efector similar a la hidrofobina de Puccinia graminis f. sp. Tritici (Pgt_Ug99_A1|8112).
(I) Estructura determinada experimentalmente de AvrSr50 QCMJC de Puccinia graminis f. sp. tritici (PDB:7MQQ).
(J) Estructura prevista del supuesto efector tipo Tin2 de Ustilago maydis (UMAG_05930).
(K) Estructura determinada experimentalmente del efector BEC1054 similar a ARNasa de Blumeria graminis f. sp. hordei (PDB: 6FMB).
(L) Estructura determinada experimentalmente de los efectores similares a ARNasa de Blumeria graminis AvrA6 (PDB:8OXH), AvrA7 (PDB:8OXL), AvrA10 (PDB:8OXK), AvrA22 (PDB:8OXJ) y AvrPM2 (PDB:8PHY).